UCSF Chimera: Software de Modelado Molecular para Visualización y Análisis de Estructuras Moleculares
Novedad de invención
Esta invención consiste en un software que facilita el modelado y la visualización interactiva de estructuras moleculares y datos relacionados.
Propuesta de valor
El modelado y la visualización de estructuras moleculares son fundamentales para obtener información sobre la función biológica y las interacciones moleculares, así como para el diseño y desarrollo de fármacos. Para satisfacer las necesidades de los investigadores, la plataforma de software extensible UCSF Chimera ha sido desarrollada por el Recurso para la Biocomputación, la Visualización y la Informática (RBVI). Este software permite la visualización en 3D y el análisis estructural de entidades moleculares, y mejora el flujo de trabajo de los investigadores con nuevas características de extensión.
Este software ofrece las siguientes ventajas:
-Amplio conjunto de funciones accesibles a través de comandos e interfaces gráficas
-Visualización de datos moleculares en todas las escalas, incluidos grandes conjuntos moleculares
-Vistas integradas de secuencia y estructura
-Producción de imágenes y películas de alta calidad para publicación y presentación
-Diseñado para adaptarse al desarrollo externo de extensiones de software
-Amplio uso para investigadores, educadores y estudiantes académicos y de la industria
Descripción de la tecnología
Quimera de UCSF ofrece visualización en 3D de estructuras moleculares y datos relacionados, que incluyen mapas de densidad, conjuntos supramoleculares, trayectorias de dinámica molecular y alineaciones de secuencias múltiples. El usuario también puede crear imágenes y animaciones para su publicación y presentación. Además de soportar la visualización del núcleo, el software está diseñado específicamente para la extensibilidad, para permitir que los desarrolladores externos incorporen nuevas funciones deseables. Las extensiones actuales incluyen Modelos Multiescala para visualizar conjuntos moleculares a gran escala como capas virales, ViewDock para visualizar orientaciones de ligandos acoplados a pantalla, Visor de volumen para visualizar mapas de densidad y Visor de múltiples alineaciones para mostrar alineaciones de secuencias, con diafonía a cualquier estructura asociada.
Algunos ejemplos incluyen:
1. Representación gráfica de la proteína tumoral p53 que se une al ADN, con ubicaciones de mutaciones asociadas al cáncer marcadas en rojo:
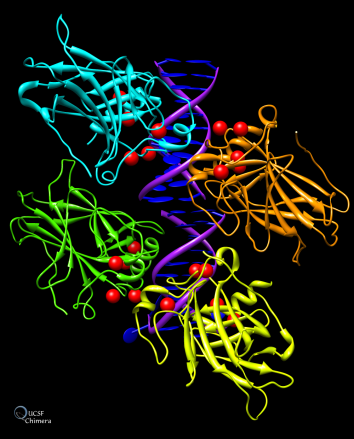
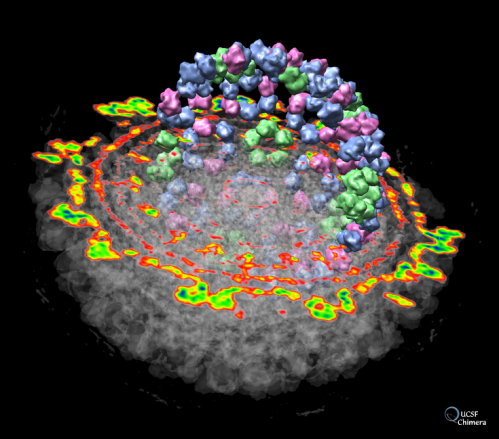
2. Representación gráfica de un mapa de densidad electrónica del virus del Bosque Semliki. El virus puede causar una encefalitis letal en roedores, pero generalmente solo síntomas leves en humanos:
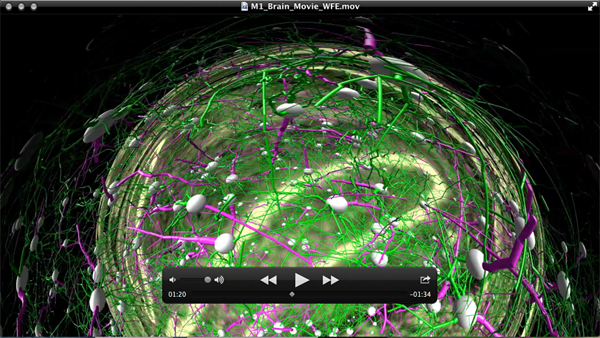
3. Video of Human Brain: Images to Atoms (en inglés). 31 de enero de 2014 (Enlace directo – https://www.youtube.com/watch?v=RE2XMzInX_o) Las imágenes aparecen distorsionadas, ya que este video estaba destinado a proyectarse en una pantalla de proyección en forma de cúpula.
Materiales relacionados
- Primera publicación: UCSF Chimera – un sistema de visualización para investigación exploratoria y análisis (J Comput Chem. 2004)
- Esta invención también tiene una lista de muchas publicaciones asociadas.