16.2: Viral Life Cycles
La via lisogenica
Un’opzione migliore per alcuni virus batterici è chiamata via lisogenica. I batteriofagi che hanno questa opzione, così come una via litica, sono noti come fago temperato. In questo percorso, il virus entra in dormienza integrandosi nel genoma ospite e rimanendo trascrizionalmente quiescente fino a quando le condizioni ambientali cambiano e riflettono una probabilità di infettare più cellule ospiti (Figura \ (\PageIndex{4}\)).
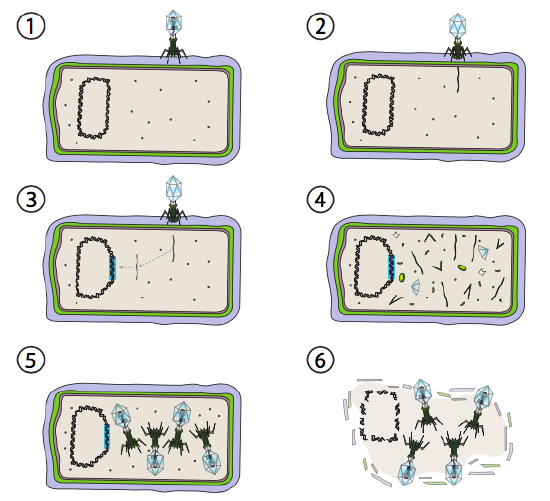
Lambda (λ) è un esempio di batteriofago temperato. Le fasi iniziali dell’infezione e dell’iniezione del genoma sono le stesse del ciclo litico, ma nelle circostanze che incoraggiano la lisogenesi, il genoma virale è integrato nel genoma ospite nel punto 3. In λ integrazione in E. coli, ciò si verifica per ricombinazione reciproca in una sequenza di coppie di 15 basi nota come sito att λ ed è facilitata dal prodotto genico Int. Finché le condizioni ambientali non favoriscono la riproduzione batterica (e quindi un numero limitato di possibili cellule ospiti), il genoma virale rimane per lo più nascosto e inattivo. L’unica eccezione significativa è un gene che codifica un repressore λ che impedisce il passo successivo e mantiene il virus dormiente.
Questo passo successivo è l’escissione del DNA del fago λ dal cromosoma ospite, e la successiva replicazione e trascrizione del DNA virale (Figura \(\PageIndex{4}\), Passo 4). Quindi, come prima, le fasi finali sono l’assemblaggio e l’accumulo di virioni e l’eventuale rottura della struttura cellulare e il rilascio delle particelle virali.
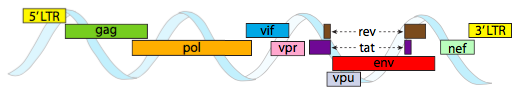
Affinché l’imballaggio nei vincoli di spazio stretti offerti dai capsidi, i genomi virali devono essere altamente economici. Ad esempio, il genoma dell’HIV (Figura \(\PageIndex{6}\)) ha diversi geni che si sovrappongono.
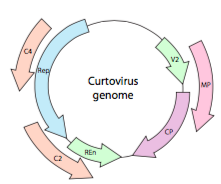
O, nel caso dei curtovirus, dei virus vegetali ssDNA (ad esempio beet curly top virus), il genoma non solo ha geni di lappatura eccessivi, ma è anche bidirezionale (Figura \(\PageIndex{7}\)) che codifica i prodotti genetici in entrambi i filamenti di DNA dopo che l’ssDNA è stato convertito in dsDNA.
Data la necessità di economia, quali geni si trovano nei virus? Uno dei più studiati del genoma virale, batteriofago lambda, contiene geni che codificano per cinque controllo trascrizionale delle proteine (che sono espressi dipende dal fatto che il fago è un lisogeni o litico modalità), una proteina che controlla il degrado di un attivatore trascrizionale, 17 proteine del capside, un excisionase che controlla l’escissione e l’inserimento del genoma del fago nel genoma ospite, un’integrazione di proteine che inserisce il genoma del fago del paese ospitante, e 3 geni che partecipano alla lisi della cellula ospite.
Il genoma dell’HIV descritto sopra è molto più piccolo di λ, a circa 9 kilobasi rispetto a 48 kb, ma ancora una volta, il tema è usare le proteine cellulari quando possibile e codificare i geni virali se necessario. Così, gag codifica per le proteine del capside, pol codifica per la trascrittasi inversa, integrasi e della proteasi dell’HIV (che fende le gag e pol prodotti del gene nel loro proteine funzionali), vif atti contro un comune della cellula ospite antivirale enzima, vpr regola l’importazione nucleare, tat fortemente aumenta la trascrizione di geni HIV, rev esportazioni di RNA virale dal nucleo, vpu è necessario per la nascita di particelle dall’host, env codifica busta virale glicoproteine, e nef promuove la sopravvivenza delle cellule infette. Le regioni LTR sono promotori molto forti per guidare l’alta espressione di questi geni.
Sebbene non sia indicato come lisogenesi, alcuni virus animali possono comportarsi in modo simile. L’esempio più importante sono i virus Baltimore Class VI-comunemente noti come retrovirus, uno dei quali è l’HIV. Il percorso di un retrovirus attraverso una cellula ospite eucariotica è illustrato di seguito (g. 5).
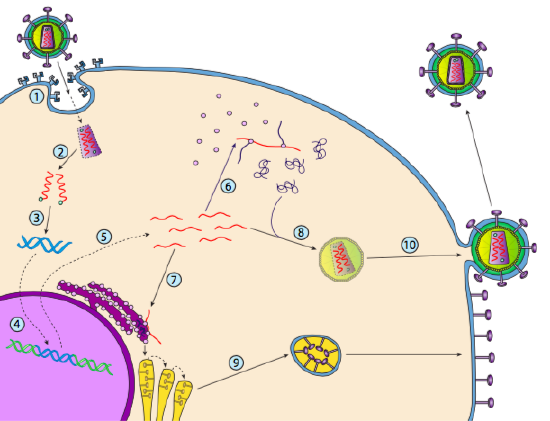
L’HIV ha un involucro, che è costellato di proteine transmembrana che sono riconosciute dalla cellula ospite, legando il virus alla superficie cellulare e avviando l’endocitosi mediata dal recettore (1). Dopo l’endocitosi, l’involucro della membrana del virione e la membrana vescicolare si fondono per rilasciare il capside e il suo contenuto (2). Dopo che il capside si dissocia nel citoplasma, i due filamenti di RNA virale vengono rilasciati insieme a una speciale polimerasi: la trascrittasi inversa, che legge un modello di RNA e sintetizza il DNA. La trascrittasi inversa utilizza anche quel nuovo DNA per sintetizzare un filamento di DNA complementare in modo che alla fine produca una versione a doppio filamento di DNA del genoma virale (3). Questo dsDNA virale viene trasportato nel nucleo dove si integra nel genoma ospite utilizzando un’altra proteina virale, integrasi
(4). Il DNA virale integrato è chiamato provirus. Il provirus può rimanere dormiente, ma se viene attivato, viene trascritto e l’RNA virale risultante viene trasportato fuori dal nucleo (5). Alcuni degli RNA virali codificano enzimi come la trascrittasi inversa e l’integrasi, o proteine del capside, che sono tutti fatti nel citoplasma (6), ma alcuni codificano le glicoproteine legate alla membrana, che vengono traslocate nell’ER (7) e infine elaborate attraverso il Golgi e incorporate nella membrana plasmatica (9). Una volta che il virione è stato assemblato (8), si lega alle proteine transmembrana virali, nucleando una “vescicola” esocitica (10) che è il virione completo di involucro virale.
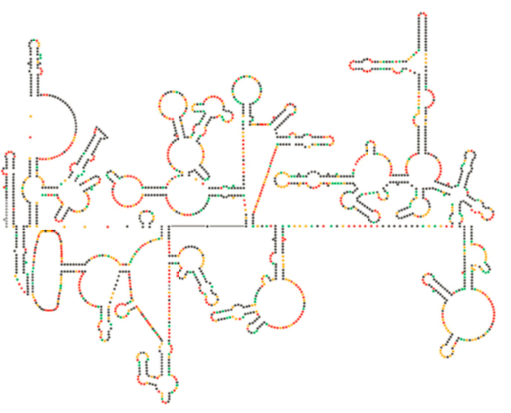
Un recente esame strutturale del genoma dell’HIV suggerisce che la struttura stessa dell’HIV RNA può svolgere un ruolo significativo nella sua propagazione all’interno delle cellule ospiti. Figura \(\PageIndex{8}\), da Watts et al, Nature 460:711-716, 2009, mostra una struttura secondaria prevista del genoma. Gli autori suggeriscono che la struttura dell’RNA in realtà può interagire con l’allungamento ribosomiale per controllare il ripiegamento delle proteine virali. Postulano anche l’estensione di questo argomento per includere importanti informazioni genetiche codificate non solo nella sequenza nucleotidica, ma la struttura secondaria e la struttura terziaria di qualsiasi virus a RNA.
Nel considerare i virus rispetto al resto di questo capitolo integrativo, ci sono due idee prioritarie da tenere a mente. In primo luogo, la sopravvivenza virale si basa sui numeri: ha bisogno di fare un numero enorme dei suoi componenti per lanciare una rete più ampia possibile per nuove cellule ospiti. Per fare questo, i promotori virali sono solitamente molto più forti dei promotori della cellula ospite, simultaneamente guidando più espressione genica virale mentre impediscono l’espressione genica ospite (dedicando le risorse cellulari alla produzione del virus). In secondo luogo, a causa dei tempi di generazione veloci, il tasso di mutazione virale e l’evoluzione è molto più veloce rispetto ai normali genomi eucariotici. Inoltre, se il virus utilizza la propria polimerasi (come la trascrittasi inversa o l’RNA replicasi), il tasso di mutazione aumenta ancora di più perché non vi è alcun controllo degli errori da parte delle polimerasi virali.