16.2: Ciclos de Vida Virales
La Vía Lisogénica
Una mejor opción para algunos virus bacterianos se llama la vía lisogénica. Los bacteriófagos que tienen esta opción, así como una vía lítica, se conocen como fagos templados. En esta vía, el virus entra en latencia al integrarse en el genoma del huésped y permanecer en reposo transcripcional hasta que las condiciones ambientales cambien y reflejen la probabilidad de que más células huésped infecten (Figura \(\pageIndex{4}\)).
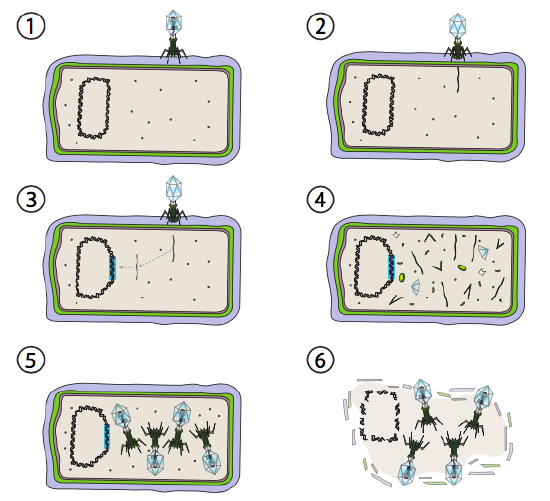
Lambda (λ) es un ejemplo de bacteriófago templado. Las etapas iniciales de la infección y la inyección del genoma son las mismas que el ciclo lítico, pero en condiciones que fomentan la lisogenia, el genoma viral se integra en el genoma huésped en el paso 3. En λ integración en E. coli, esto ocurre por recombinación recíproca en una secuencia de 15 pares de bases conocida como el sitio att λ y es facilitado por el producto del gen Int. Mientras las condiciones ambientales no sean propicias para la reproducción bacteriana (y por lo tanto un número limitado de posibles células huésped), el genoma viral permanece mayormente oculto e inactivo. La única excepción significativa es un gen que codifica un represor λ que previene el siguiente paso y mantiene al virus inactivo.
El siguiente paso es la escisión del ADN del fago λ del cromosoma huésped, y la posterior replicación y transcripción del ADN viral (Figura \(\pageIndex{4}\), Paso 4). Luego, como antes, los pasos finales son el ensamblaje y la acumulación de viriones, y la eventual ruptura de la estructura celular y la liberación de las partículas virales.
Para poder empaquetarse en las restricciones de espacio reducidas que ofrecen los capsidos, los genomas virales deben ser altamente económicos. Por ejemplo, el genoma del VIH (Figura \(\pageIndex{6}\)) tiene varios genes que se superponen.
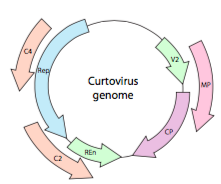
O, en el caso de los curtovirus, los virus de las plantas de ADNss (por ejemplo, el virus de la parte superior rizada de la remolacha), el genoma no solo tiene genes de traslapado de repeticiones excesivas, sino que incluso es bidireccional (Figura \(\pageIndex{7}\)) codificando productos genéticos en ambas hebras de ADN después de que el ADNss se haya convertido en adNSD.
Dada la necesidad de economía, ¿qué genes se encuentran en los virus? Uno de los genomas virales más estudiados, el bacteriófago λ, contiene genes que codifican cinco proteínas de control transcripcional (las cuales se expresan depende de si el fago está en modo lisogénico o lítico), una proteína de unión que controla la degradación de un activador transcripcional, 17 proteínas de la cápside, una escisionasa que controla la escisión e inserción del genoma del fago en el genoma del huésped, una proteína de integración que inserta el genoma del fago en el del huésped y 3 genes que participan en la lisis de la célula huésped.
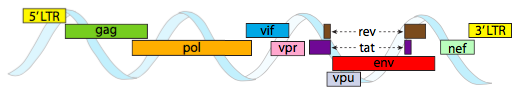
El genoma del VIH descrito anteriormente es mucho más pequeño que λ, alrededor de 9 kilobases en comparación con 48 kb, pero de nuevo, el tema es usar proteínas celulares cuando sea posible y codificar genes virales si es necesario. Por lo tanto, gag codifica las proteínas de la cápside, pol codifica la transcriptasa inversa, la integrasa y la proteasa del VIH (que escinde los productos de los genes gag y pol en sus proteínas funcionales), vif actúa contra una enzima antiviral común de la célula huésped, vpr regula la importación nuclear, tat aumenta fuertemente la transcripción de los genes del VIH, rev exporta ARN viral desde el núcleo, vpu es necesaria para la formación de partículas del huésped, env codifica las glicoproteínas de la envoltura viral y nef promueve la supervivencia de las células infectadas. Las regiones LTR son promotores muy fuertes para impulsar la alta expresión de estos genes.
Aunque no se conoce como lisogenia, algunos virus animales pueden comportarse de manera similar. El ejemplo más destacado son los virus de Clase VI de Baltimore, comúnmente conocidos como retrovirus, uno de los cuales es el VIH. La trayectoria de un retrovirus a través de una célula huésped eucariota se muestra a continuación ( g. 5).
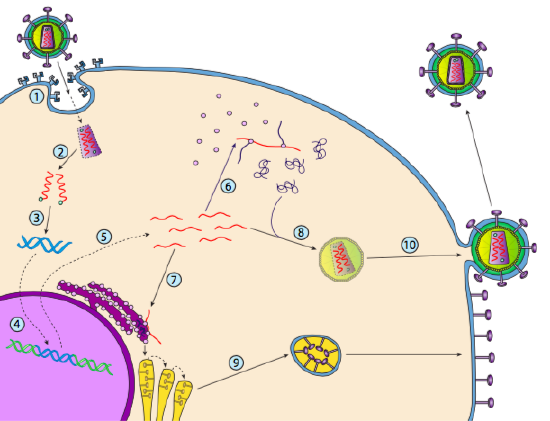
El VIH tiene una envoltura, que está repleta de proteínas transmembranas que son reconocidas por la célula huésped, uniendo el virus a la superficie celular e iniciando la endocitosis mediada por receptores (1). Después de la endocitosis, la envoltura de membrana del virión y la membrana vesicular se fusionan para liberar la cápside y su contenido (2). Después de que la cápside se disocia en el citoplasma, las dos hebras de ARN viral se liberan junto con una polimerasa especial: la transcriptasa inversa, que lee una plantilla de ARN y sintetiza el ADN. La transcriptasa inversa también utiliza ese nuevo ADN para sintetizar una cadena de ADN complementaria para que finalmente produzca una versión de ADN de doble cadena del genoma viral (3). Este dsDNA viral se transporta al núcleo, donde se integra en el genoma del huésped utilizando otra proteína viral, la integrasa
(4). El ADN viral integrado se llama provirus. El provirus puede permanecer latente, pero si se activa, se transcribe y el ARN viral resultante se transporta fuera del núcleo (5). Algunos de los ARN virales codifican enzimas como la transcriptasa inversa y la integrasa, o proteínas de la cápside, todas las cuales se producen en el citoplasma (6), pero algunas codifican glicoproteínas unidas a la membrana, que se translocan en la sala de Emergencias (7) y finalmente se procesan a través del Golgi e incorporan a la membrana plasmática (9). Una vez que el virión ha sido ensamblado (8), se une a las proteínas transmembranas virales, nucleando una «vesícula» exocítica (10) que es el virión completo con envoltura viral.
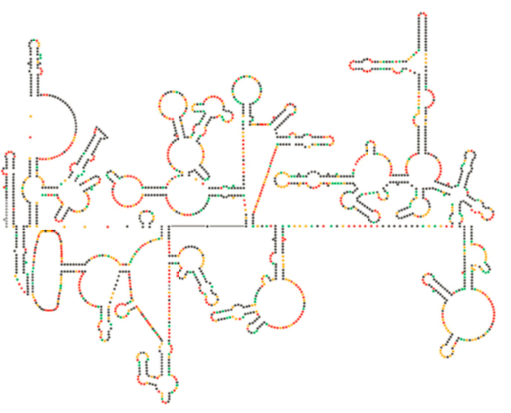
El reciente examen estructural del genoma del VIH sugiere que la estructura del ARN del VIH en sí puede desempeñar un papel importante en su propagación dentro de las células huésped. La figura \(\pageIndex{8}\), de Watts et al, Nature 460:711-716, 2009, muestra una estructura secundaria predicha del genoma. Los autores sugieren que la estructura del ARN en realidad puede interactuar con el alargamiento ribosomal para controlar el plegamiento de las proteínas virales. También postulan la extensión de este argumento para incluir información genética importante codificada no solo en la secuencia de nucleótidos, sino en la estructura secundaria y terciaria de cualquier virus ARN.
Al considerar los virus con respecto al resto de este capítulo integrador, hay dos ideas primordiales a tener en cuenta. En primer lugar, la supervivencia viral se basa en números: necesita hacer un gran número de sus componentes para lanzar una red lo más amplia posible para nuevas células huésped. Para hacer esto, los promotores virales son generalmente mucho más fuertes que los promotores de la célula huésped, al mismo tiempo que impulsan una mayor expresión génica viral y previenen la expresión génica del huésped (al dedicar recursos celulares a la producción de virus). En segundo lugar, debido a los tiempos de generación rápidos, la tasa de mutación y evolución viral es mucho más rápida que los genomas eucariotas normales. Además, si el virus utiliza su propia polimerasa (como la transcriptasa inversa o la ARN replicasa), la tasa de mutación aumenta aún más porque no hay verificación de errores por parte de las polimerasas virales.